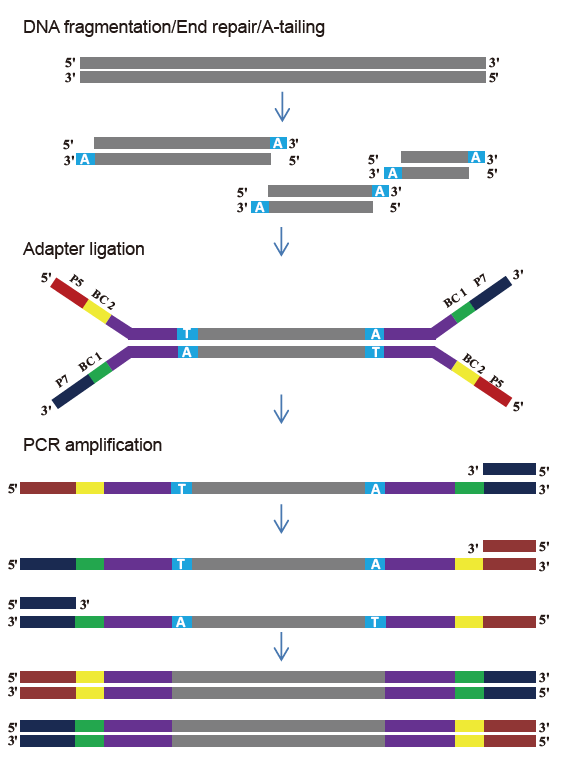
Biblioteko TIANSeq DirectFast (illumina)
Trajtoj
■ Bona sekvenca unuformeco: Neniu baza antaŭjuĝo de la DNA-fragmentiĝa procezo kaj PCR-plifortiga procezo.
■ Alta biblioteka konverta efikeco: la alta efikeca biblioteka konstruo povas esti certigita por 1 ng-DNA-specimenoj.
■ Rapida funkciado: La tuta procezo de konstruado de biblioteko bezonas nur 2,5 horojn.
■ Kostefika: Ne necesas specialaj instrumentoj kaj ekipaĵoj。
Specifo
Tipo: DNA-biblioteko-preparo por iluminina altrapida sekvenca platformo
Specimeno: Genoma DNA aŭ granda fragmenta DNA
Celo: Duoble-senhelpa DNA
Komenca ekzempla enigo: 1 ng- 1 μg
Tempo de operacio: 2,5 horo
Laŭfluaj aplikoj: Sekvencado sur iluminina platformo
Ĉiuj produktoj povas esti personecigitaj por ODM / OEM. Por detaloj,bonvolu alklaki Agorditan Servon (ODM / OEM)
Fleksebla specimena enigo kaj fragmenta grandeco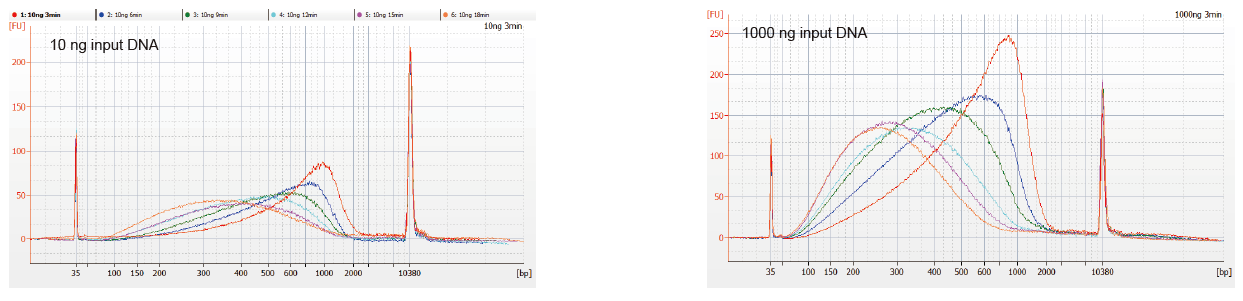 |
Figuro 1. DNA-fragmentiĝaj profiloj de malsama reaga tempo. 10 ng kaj 1000 ng DNA estis fragmentigitaj per TIANSeq DirectFast DNA Library Kit. La reagproduktoj traktitaj kun malsama reagtempo estis purigitaj per 1,8 × Ampure XP-magnetaj artperloj kaj analizitaj fare de Angilent 2100. |
Covaris-Kiel Sekvenca Priraportado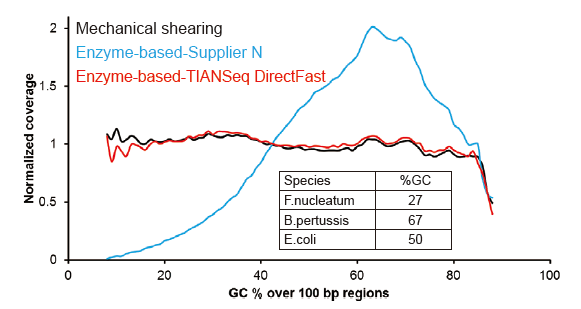 |
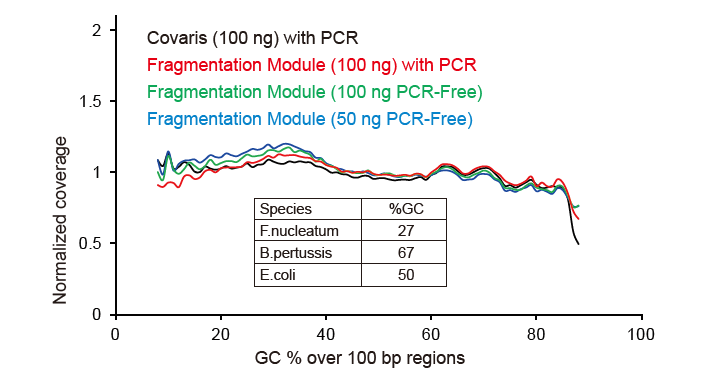
Figuro 2. Komparo de genara priraportado de malsamaj bibliotekaj preparaj metodoj. Tri bakteriaj genomic-DNA kun malsama GC-enhavo estas miksitaj ekimolare, kaj sekvenca genoma priraportado rezultas de 100 ng de miksitaj DNA-bibliotekoj uzantaj ĉi tiujn metodojn. La rezultoj montras, ke la TIANSeq DirectFast Library Kit havas la saman efikon al fragmentiĝo de DNA kiel mekanika tondado, kaj ne ekzistas baza antaŭjuĝo por fragmentiĝo. |
Neniu Sistema Biaso Por Malalte Kiel 1 ng Eniga DNA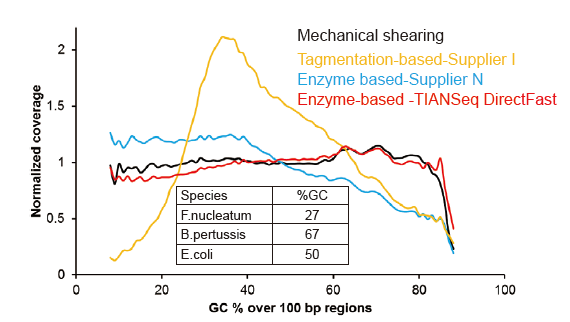 |
Figuro 3. Komparo de genara priraportado de malsamaj bibliotekaj preparaj metodoj. Tri bakteriaj genomic-DNA kun malsama GC-enhavo estas miksitaj ekimolare, kaj sekvenca genoma priraportadrezulto de 1 ng de miksitaj DNA-bibliotekoj uzantaj ĉi tiujn metodojn estis komparita. La rezultoj montras, ke la TIANSeq DirectFast Library Kit havas konsekvencan fragmentigan efikon kun la mekanika tondado eĉ por DNA-enigo tiel malmulte kiel 1 ng, kaj ne ekzistas baza antaŭjuĝo. |
| Kapabla PCR-Libera Laborfluo
|
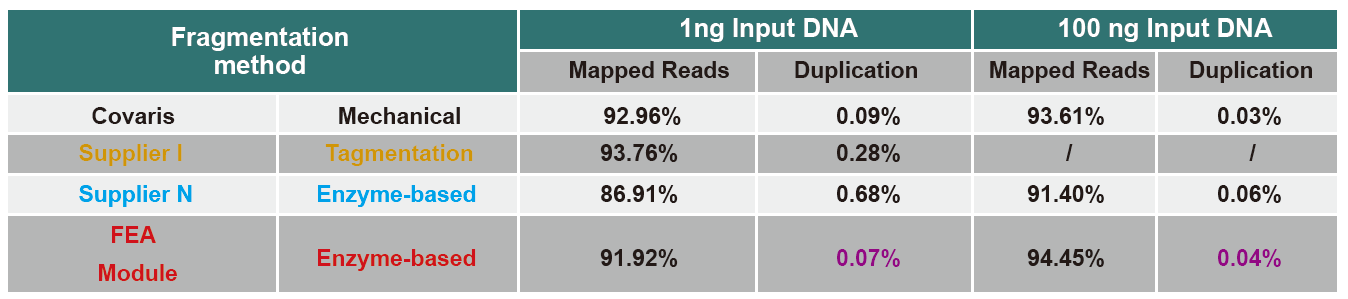
Figuro 4. Malsamaj enigoj de genomika DNA estis uzataj por konstrui la bibliotekon per PCR aŭ senpaga biblioteka konstruado, kaj la rezultoj pri kovrado de genaro estis komparitaj. La rezultoj montras, ke kun la unu-tuba funkciado kaj efikaj bibliotekaj konstruaj paŝoj, la DNA-biblioteko konstruita per TIANSeq DirectFast Library Kit konservas altan konsekvencon kun la mekanika tondado en fragmenta sinsekva kovra distribuo por ambaŭ PCR-riĉigo sen PCR-laborfluo. |
Statistiko de Biblioteka Konstrua Efikeco kaj Rendimento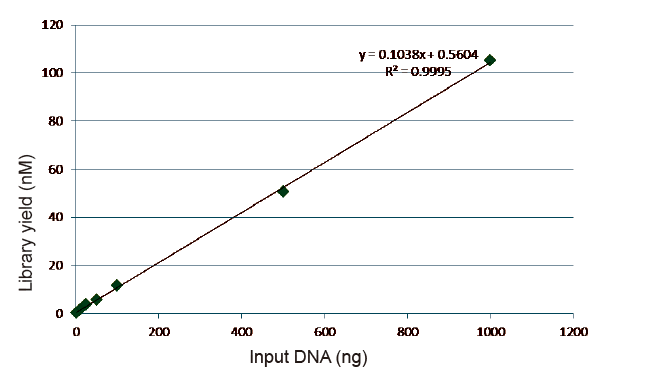 |
Figuro 5. Rezultoj de kvanta analizo de biblioteka DNA akirita de qPCR post konstruado de biblioteko per PCR-libera metodo por specimenoj kun malsamaj komencaj kvantoj (1, 10, 25, 50, 100, 500,1000 ng). Linia regresa analizo montras, ke la biblioteka rendimento havas bonan linian rilaton en larĝa specimena eniga gamo. Por DNA-enigo tiel malmulte kiel 1 ng, la efikeco de biblioteka konstruado ne malpliiĝas. |
Komparo de Sekvencaj Datumoj de Malsamaj Produktoj
Nuntempe altrapida sinsekva teknologio ĉefe baziĝas sur sekvenca generacia sinsekva teknologio. Ĉar la lega longo de sekva generacia sinsekva teknologio estas limigita, ni devas rompi la plenan longan sinsekvon en malgrandajn fragmentajn bibliotekojn por sinsekvi. Laŭ la bezonoj de malsamaj sekvencaj eksperimentoj, ni kutime elektas unu-finitan sekvencadon aŭ duoble-finitan sekvencadon. Nuntempe la DNA-fragmentoj de la sekvenca generacia biblioteko estas ĝenerale distribuitaj en la gamo de 200-800 bp.

a) DNA estas malbonkvalita kaj enhavas inhibitorojn. Uzu altkvalitajn DNA-specimenojn por eviti inhibicion de enzima agado.
b) La kvanto de DNA-specimeno estas nesufiĉa kiam oni uzas PCR-liberan metodon por konstrui DNA-bibliotekon. Kiam la enigo de la fragmenta DNA superas 50 ng, senpaga laborfluo povas esti selekte efektivigita dum la konstrua procezo de biblioteko. Se la kopia nombro de la biblioteko estas tro malalta por esti rekte vicigita, la DNA-biblioteko povas esti plifortigita per PCR post la adaptila ligado.
c) RNA-poluado kondukas al malpreciza komenca DNA-kvantigado. RNA povas forigi per traktado kun RNase.
A-1
a) Malgrandaj fragmentoj (60 bp-120 bp) aperas Malgrandaj fragmentoj estas kutime adaptilaj fragmentoj aŭ dimeroj formitaj de adaptiloj. Purigado per magnetaj artperloj Agencourt AMPure XP povas efike forigi ĉi tiujn adaptilajn fragmentojn kaj certigi sekvencan kvaliton.
b) Grandaj fragmentoj aperas en la biblioteko post PCR-plifortigo. La grandeco de la DNA-fragmento de la biblioteko pliiĝos je 120 bp post kiam la adaptilo estas ligita. Se la DNA-fragmento pliiĝas je pli ol 120 bp post la adaptilligo, ĝi povus esti kaŭzita de nenormala fragmenta plifortigo de troa PCR-plifortigo. Redukti la nombron de PCR-cikloj povas malhelpi la situacion.
c) Nenormala grandeco de bibliotekaj DNA-fragmentoj post ligilo de adaptilo La longo de la adaptilo en ĉi tiu ilaro estas 60 bp. Kiam la du finoj de la fragmento estas ligitaj al la adaptiloj, la longo nur kreskos je 120 bp. Kiam vi uzas alian adaptilon krom tiu provizita de ĉi tiu ilaro, bonvolu kontakti la provizanton por doni gravajn informojn kiel ekzemple adaptila longo. Bonvolu certigi, ke la eksperimenta laborfluo kaj funkciado sekvas la paŝojn priskribitajn en la manlibro.
d) Nenormala DNA-fragmenta grandeco antaŭ la adaptila ligado La kialo de ĉi tiu problemo povas esti kaŭzita de malĝustaj reagaj kondiĉoj dum DNA-fragmentiĝo. Malsamaj reagaj tempoj devas esti uzataj por malsama DNA-enigo. Se la enigaĵo de DNA estas pli ol 10 ng, ni rekomendas elekti la reagan tempon de 12 min kiel komencan tempon por optimumigo, kaj la grandeco de fragmento produktita tiutempe estas ĉefe inter 300-500 bp. Uzantoj povas pliigi aŭ malpliigi la longon de DNA-fragmentoj por 2-4 min laŭ siaj propraj postuloj por optimumigi la DNA-fragmentojn kun la bezonata grandeco.
A-2
a) Fragmenta tempo ne estas optimumigita Se la fragmenta DNA estas tro malgranda aŭ tro granda, bonvolu raporti al la Gvidlinioj por Fragmenta Tempo-Selektado provizita en la instrukcio por determini la reagan tempon, kaj uzi ĉi tiun tempopunkton kiel kontrolon, aldone starigu reakcia sistemo plilongigi aŭ mallongigi 3 min por fari pli precizan ĝustigon dum fragmentiĝa tempo.
A-3
Nenormala grandeca distribuado de DNA post fragmentiĝa traktado
a) Malĝusta degela metodo de fragmentiĝa reakciilo, aŭ la reakciilo ne estas tute miksita post degelo. Degelu la reakciilon 5 × Fragmentation Enzyme Mix sur glacio. Post degelo, miksu la reakciilon egale milde frapetante la fundon de la tubo. Ne vortumu la reakciilon!
b) La DNA-eniga specimeno enhavas EDTA aŭ aliajn malpurigaĵojn Malplenigo de salaj jonoj kaj kelataj agentoj en la DNA-puriga paŝo estas aparte grava por la sukceso de la eksperimento. Se DNA solviĝas en 1 × TE, uzu la metodon provizitan en la instrukcio por fari fragmentiĝon. Se la EDTA-koncentriĝo en la solvaĵo estas necerta, oni rekomendas purigi la DNA kaj solvi ĝin en dejonigita akvo por posta reago.
c) Malĝusta komenca DNA-kvantigado La grandeco de fragmenta DNA estas proksime rilata al la kvanto de enigaĵo de DNA. Antaŭ fragmentiĝa traktado, preciza kvantigo de DNA per Qubit, Picogreen kaj aliaj metodoj estas esenca por determini la ĝustan kvanton de DNA en la reakcia sistemo.
d) La preparado de la reaga sistemo ne sekvas la instrukcion. La preparado de fragmenta reaga sistemo devas esti farita sur glacio strikte laŭ la instrukcioj. Por certigi la plej bonan efikon, ĉiuj reagokomponentoj devas esti metitaj sur glacion kaj la preparado de reagsistemo devas esti aranĝita post kompleta malvarmigo. Post kiam la preparo finiĝos, bonvolu fliki aŭ pipeti por miksi ĝisfunde. Ne vorticu!
1. Nedeca miksa metodo (vortico, perforta oscilado ktp.) Kaŭzos nenormalan distribuadon de bibliotekaj fragmentoj (kiel montrite en la sekva figuro), tiel influante la kvaliton de la biblioteko. Tial, kiam vi preparas la reagan solvon de Fragmentiga Miksaĵo, bonvolu milde pipeti supren kaj malsupren por miksi, aŭ uzu la fingropinton por frapi kaj miksi egale. Atentu ne miksi kun vortico.

2. Alta pureca DNA devas esti uzata por konstruado de biblioteko
■ Bona DNA-integreco: la elektroforeza bendo estas pli ol 30 kb, sen postsekvado
■ OD260 / 230:> 1.5
■ OD260 / 280: 1.7-1.9
3. Eniga kvanto de DNA devas esti ĝusta. Oni sugestas uzi Qubit kaj PicoGreen-metodojn por kvantigi DNA, anstataŭ Nanodrop.
4. La enhavo de EDTA en DNA-solvo devas esti determinita. EDTA havas grandan influon sur la fragmentiĝa reago. Se la enhavo de EDTA estas alta, DNA-purigado devas esti farita antaŭ la posta testo.
5. La fragmenta reaga solvo devas esti preparita sur glacio. La fragmenta procezo estas sentema al reaga temperaturo kaj tempo (precipe post aldono de amplifilo). Por certigi la precizecon de reaga tempo, bonvolu prepari reagsistemon sur glacio.
6. La reaga tempo de fragmentiĝo devas esti ĝusta La reaga tempo de la fragmenta paŝo rekte influos la grandecon de la fragmentaj produktoj, tiel influos la grandecan distribuadon de DNA-fragmentoj en la biblioteko.
1. Kia specimeno aplikeblas al ĉi tiu ilaro?
La aplikebla specimeno de ĉi tiu ilaro povas esti totala RNA aŭ purigita mRNA kun bona RNA-integreco. Se totala RNA estas uzata por konstrui la bibliotekon, oni rekomendas uzi la rRNA-malplenigan ilaron (Cat # 4992363/4992364/4992391) por unue forigi rRNA.
2. Ĉu FFPE-specimenoj povas esti uzataj por konstrui bibliotekon per ĉi tiu ilaro?
La mRNA en specimenoj de FFPE iom difektiĝos, kun relative malbona integreco. Kiam vi uzas ĉi tiun ilaron por la biblioteka konstruado, oni rekomendas optimumigi la fragmentan tempon (mallongigi la fragmentan tempon aŭ ne plenumi fragmentiĝon).
3. Uzante la grandecan elekton-paŝon provizitan en la produkta manlibro, kio povus kaŭzi la enmetitan segmenton aperi iomete?
Elektado de grandeco devas plenumi strikte konforme al la paŝo de grandeco en ĉi tiu produkta manlibro. Se estas devio, la kialo povus esti, ke la magnetaj bidoj ne balanciĝas al ĉambra temperaturo aŭ ne estas tute miksitaj, la pipeto ne estas preciza aŭ la likvaĵo restas en la pinto. Oni rekomendas uzi la konsiletojn kun malalta adsorbado por la eksperimento.
4. Elekto de adaptiloj en biblioteka konstruado
La biblioteka konstrukompleto ne enhavas adaptilan reakciilon, kaj oni rekomendas uzi ĉi tiun ilaron kune kun TIANSeq-Ununura Indeksa Adaptilo (Illumina) (4992641/4992642/4992378).
5. QC de la biblioteko
Biblioteka kvanta detekto: Qubit kaj qPCR estas uzataj por determini la amasan koncentriĝon kaj molan koncentriĝon de la biblioteko respektive. La operacio strikte konformas al la produkta manlibro. La koncentriĝo de la biblioteko ĝenerale plenumos la postulojn de NGS-sinsekvado. Malkaŝo de biblioteka distribua gamo: Uzante Agilent 2100 Bioanalyzer por detekti la bibliotekan distribuan gamon.
6. Elekto de plifortiga cikla nombro
Laŭ la instrukcioj, la nombro de PCR-cikloj estas 6-12, kaj la nombro de necesaj PCR-cikloj devas esti elektita laŭ la specimena enigo. En alt-rendimentaj bibliotekoj, super plifortigo kutime okazas en diversaj gradoj, kiu manifestiĝas per iomete pli granda pinto post la pinto de la celintervalo en la detekto de Agilent 2100 Bioanalyzer, aŭ la detektita koncentriĝo de Qubit estas pli malalta ol tiu de qPCR. Milda super plifortigo estas normala fenomeno, kiu ne influas bibliotekan sinsekvadon kaj postan datuman analizon.
7. Pikiloj aperas en la detekta profilo de Agilent 2100 Bioanalyzer
La apero de pikiloj en detekto de Bioanalizilo Agilent 2100 estas pro la neegala fragmentiĝo de specimenoj, kie estos pli da fragmentoj en certa grandeco, kaj ĉi tio fariĝos pli evidenta post PCR-riĉigo. Ĉi-kaze oni sugestas ne plenumi la elekton de grandeco, do starigu la fragmentan kondiĉon al 94 ° C dum 15 min kovataj, kie la fragmenta distribuo estas malgranda kaj koncentrita, kaj la homogeneco povas esti plibonigita.
Produktaj kategorioj
KIAL ELEKTU NIN
Ekde ĝia estiĝo, nia fabriko disvolvas unuajn mondklasajn produktojn konforme al la principo
de kvalito unue. Niaj produktoj akiris bonegan reputacion en la industrio kaj valoran fidon ĉe novaj kaj malnovaj klientoj ..