TIANScriptⅡ RT-Ilaro
Trajtoj
■ Alta enzima agado kaj efikeco: Alta inversa transkriptasa agado kaj bona kongruo en postaj eksperimentoj.
■ Larĝa substrata gamo: Taŭga por ĉiuj RNA, precipe RNA-ŝablonoj kun kompleksaj duarangaj strukturoj.
■ Longa RT-longo: La sintezo de la cDNA-unua fadeno povas atingi 12 kb.
■ Simpla funkciado: Simple aldonu la bezonatajn erojn en unu paŝo sen aldoni reakciilon dum la operacio.
Aplikoj
■ Sintezo de cDNA-unua fadeno.
■ Konstruado de cDNA-biblioteko.
■ Unupaŝa RT-PCR.
■ RACE-analizo.
Ĉiuj produktoj povas esti personecigitaj por ODM / OEM. Por detaloj,bonvolu alklaki Agorditan Servon (ODM / OEM)
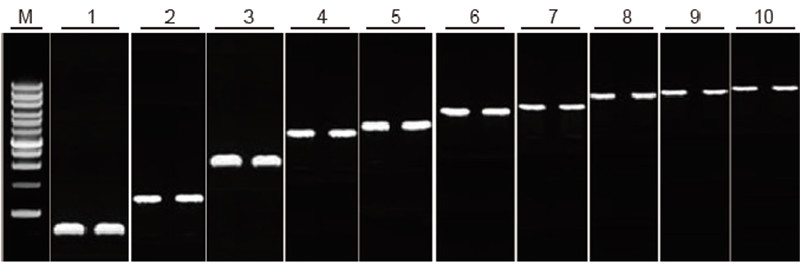 |
Inversa transskriba kapablo de TIANScript II RT-Ilaro por malsamaj longaj fragmentoj Metodo: Inversa transskribo: Konsultu la instruan manlibron de TIANScript II RT Kit. Rezultoj: La ĝela bildo montras la amplifajn rezultojn de 10 celaj genoj kun malsamaj longoj post inversa transskribo de 1 μg de totala RNA. 2 μl de inversaj transskribaj produktoj estis ŝarĝitaj po unu vojo. Plifortiga sistemo (PCR): 20 μl; Specimena ŝarĝo: 5 μl; Markilo: D15000 + 1 kb DNA-Ŝtupetaro; Koncentriĝo de ĝelo: 1%; Kondiĉoj de elektroforezo: 6 V / cm, 20 min Diagramo de ĉiu leno: M: DNA-Markilo; 1: Produkta longo: 120 bp; 2: Produkta longo: 1 kb; 3: Produkta longo: 2,5 kb; 4: Produkta longo: 3,2 kb; 5: Produkto longo: 4,6 kb; 6: Produkta longo: 6,8 kb; 7: Produkta longo: 7,6 kb; 8: Produkta longo: 8,9 kb; 9: Produkta longo: 10 kb; 10: Produkta longo: 12 kb; |
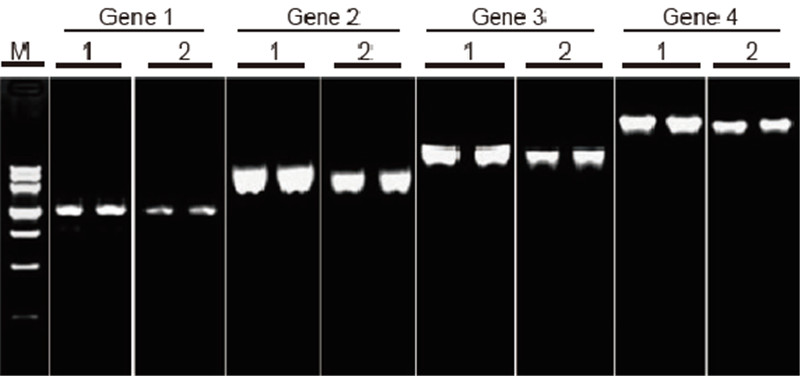 |
Komparo de efikeco kaj specifeco de TIANScript II RT Kit kaj produktoj de aliaj provizantoj en inversa transskribo de longaj ŝablonoj Materialoj: Totala RNA de homaj aliĝantaj ĉeloj. RT-PCR-komenca kvanto: 2 μl-inversa transskriba produkto (50 ng / μl) Metodo: Inversa transskribo: Vidu la instrukcion de TIANScript II RT Kit. Rezultoj: La ĝela bildo montras la amplifajn rezultojn de 6 celaj genoj kun malsamaj longoj post inversa transskribo de 1 μg totala RNA de homaj aliĝantaj ĉeloj uzantaj M-MLV de Provizanto A kaj TIANGEN TIANScript II RT-Ilaro. Plifortiga sistemo (PCR): 20 μl; Specimena ŝarĝo: 5 μl; Markilo: DNA MarkerIII; Koncentriĝo de ĝelo: 1%; Kondiĉoj de elektroforezo: 6 V / cm, 20 min. Diagramo de ĉiu leno: M: DNA-Markilo; 1: Plifortigaj rezultoj de la cDNA-inversa transskribo per TIANScriptII RT Kit; 2. Plifortigaj rezultoj de la cDNA-inversa transskribo uzante koncernan produkton de Provizanto A. La longo de produkto de geno 1 estas 1,3 kb; La longo de geno 2-produkto estas 3,0kb; La longo de geno 3-produkto estas 5,0 kb; La longo de geno 4-produkto estas 7,5 kb. |
A-1-RNA estas degradita
——Purigi altkvalitan RNA sen poluado. La materialo el kiu eltiras RNA devas esti kiel eble plej freŝa por eviti degradiĝon de RNA. Analizu RNA-integrecon sur denaturigita ĝelo antaŭ RT-reago. Post eltiro de RNA, ĝi devas esti konservita en 100% formamido. Se RNase-inhibilo estas uzata, la hejtotemperaturo devas esti <45 ° C, kaj la pH devas esti malpli ol 8,0, alie la inhibilo liberigos ĉiun ligitan RNase. Cetere, RNase-inhibilo devas esti aldonita en solvoj enhavantaj ≥ 0,8 mM TDT.
A-2 RNA enhavas inhibitorojn de inversaj transskribaj reagoj
—— Inversaj transskribaj inhibitoroj inkluzivas SDS, EDTA, glicerinon, natrian pirofosfaton, spermidinon, formamidon, guanidinan salon, ktp. Miksu la kontrolan RNA kun la specimeno, kaj komparu la rendimenton kun la kontrola RNA-reago por kontroli ĉu ekzistas inhibilo. Lavu RNA-precipitaĵon kun 70% (v / v) etanolo por forigi inhibitorojn.
A-3 Nesufiĉa kalcinado de enkondukoj uzitaj por sintezi la unuan fadenon de cDNA
——Determini, ke la temperado de kalcinado taŭgas por la enkondukoj uzataj en la eksperimento. Por hazardaj heksameroj, oni rekomendas teni la temperaturon je 25 ° C dum 10 minutoj antaŭ ol atingi la reagan temperaturon. Por genospecifaj enkondukoj (GSP), provu alian GSP, aŭ ŝanĝu al oligo (dT) aŭ hazarda heksamero.
A-4 Malgranda kvanto de starta RNA
——Pliigi la kvanton de RNA. Por RNA-specimenoj malpli ol 50 ng, 0,1 μg al 0,5 μg de acetilo BSA povas esti uzata en la unua fadena cDNA-sintezo
A-5 La cela sekvenco ne estas esprimita en la analizitaj ŝtofoj.
—— Provu aliajn ŝtofojn.
A-6-PCR-reago malsukcesas
——Por du-ŝtupa RT-PCR, la cDNA-ŝablono en la PCR-ŝtupo ne povas superi 1/5 de la reaga volumo.
A-1 Nespecifa kalcado de enkondukoj kaj ŝablonoj
——La 3'-fino de enkondukoj ne devas enhavi 2-3 dG aŭ dC. Uzu Gen-specifajn enkondukojn en la unua fadena sintezo anstataŭ hazardaj enkondukoj aŭ oligo (dT). Uzu pli altan temperadon en la unuaj cikloj, kaj poste pli malaltan temperaturon. Uzu varma-komencan Taq-DNA-polimerazon por PCR por plibonigi la specifecon de la reago.
A-2 Malbona projektado de genospecifaj enkondukoj
—— Sekvu la samajn principojn por plifortiga enkonduka projektado.
A-3-RNA poluita per genomika DNA
——Trakti RNA-on kun PCR-grado DNase I. Starigu kontrolan reagon sen inversa transskribo por detekti poluadon de DNA.
A-4 Formado de enkonduka dimero
——Desegni enkondukojn sen komplementaj sekvencoj ĉe la 3 'fino.
A-5 Tro alta Mg2+ koncentriĝo
——Optimigi Mg2+ koncentriĝo por ĉiu ŝablono kaj enkonduka kombinaĵo
A-6 Poluita per fremda DNA
——Uzu aerosol-rezistajn konsiletojn kaj UDG-enzimojn.
A-1 La enhavo de la unua fadena produkto estas tro alta
——Redukti la kvanton de la unua fadena produkto en la konvencia PCR-reaga paŝo.
A-2 Tro alta enkonduka kvanto en PCR-reago
——Redukti enkondukan enigon.
A-3 Tro multaj cikloj
——Optimigi reagajn kondiĉojn de PCR kaj redukti nombron de ciklo de PCR.
A-4 Tro malalta temperado
—— Pliigi kalcinan temperaturon por malebligi nespecifajn komencon kaj etendon.
A-5 Nespecifa plifortigo de oligonucleotidaj fragmentoj generitaj de DNase-degenero de DNA — —Ekstrakti altkvalitan RNA por malebligi poluadon de DNA.
RT-PCR devas inversigi transskribi RNA en cDNA, kaj tiam uzi la inversan transskribitan cDNA kiel ŝablonon por PCR-reago por plifortigi la celan fragmenton. Elektu aŭ hazardajn enkondukojn, Oligo dT kaj genajn specifajn enkondukojn laŭ la specifaj kondiĉoj de la eksperimento. Ĉiuj supraj enkondukoj povas esti uzataj por mallonga eŭkariota ĉela mRNA sen harpingla strukturo.
Hazarda enkonduko: Taŭga por longa RNA kun harpingla strukturo, same kiel ĉiaj RNA kiel rRNA, mRNA, tRNA, ktp. Ili estas ĉefe uzataj por RT-PCR-reago de unu ŝablono.
Oligo dT: Taŭga por RNA kun PolyA-vosto (prokariota RNA, eŭkariota Oligo dT-rRNA kaj tRNA ne havas PolyA-vostojn). Ĉar Oligo-dT estas ligita al PolyA-vosto, la kvalito de RNA-specimenoj devas esti alta, kaj eĉ malgranda kvanto de degenero multe reduktos la kvanton de plenlonga cDNA-sintezo.
Gen-specifa enkonduko: Komplementa al la ŝablona vico, taŭga por situacioj, kie la cela vico estas konata.
Estas du manieroj:
1. Interna referenca metodo: Teorie, cDNA estas DNA-fragmentoj de malsamaj longoj, do la rezulto de elektroforezo estas ŝmirmakulo. Se RNA-abundo estas malalta, neniu produkto aperos en elektroforezo, sed tio ne signifas, ke neniu produkto plifortiĝos per PCR. Ĝenerale, interna referenco povas esti uzata por detekti cDNA. Se la interna referenco havas rezultojn, la kvalito de cDNA povas esti esence garantiita (en kelkaj kazoj, se la cela gena fragmento estas tro longa, povas esti esceptoj).
2. Se estas konata geno plifortigita per ĉi tiu ŝablono, ĝi povas esti kontrolita per la enkondukoj de ĉi tiu geno. La plifortigo de interna referenco ne nepre signifas, ke ne ekzistas problemo kun cDNA. Ĉar interna referenco havas grandan abundon en cDNA, ĝi estas facile plifortigebla. Se cDNA parte malpliiĝas pro diversaj kialoj, laŭ la perspektivo de probablo, PCR-rezultoj de malaltaj abundaj celaj genoj multe influos. Dum interna referenco ankoraŭ abundas, la plifortigo probable ne influos.
Parte degradas RNA. Detekti la integrecon kaj purigi RNA
La enhavoj de RNA de malsamaj specioj povas esti malsamaj, sed ĝenerale, la ĉerpita totala RNA devas enhavi du klarajn 28S kaj 18S-bendojn en ĝela elektroforezo, kaj la brilo de la antaŭa bando devas esti duoble pli alta ol tiu de ĉi-lasta. La 5S-bendo indikas, ke RNA estis degradita, kaj ĝia brilo estas proporcia al la grado de degradado. La sukcesa plifortigo de interna referenco ne signifas, ke ne ekzistas problemo kun RNA, ĉar la interna referenco multe abundas, RNA povas esti plifortigita tiel longe kiel la degenero ne estas severa. La OD260/ OD280rilatumo de pura RNA mezurita per spektrofotometro devas esti inter 1,9 kaj 2,1. Malgranda kvanto de proteina malpureco en RNA reduktos la rilaton. Dum la valoro ne estas tro malalta, RT ne influos. Kio plej gravas por RT estas RNA-integreco.
La etendo de la interna referenca geno povas nur indiki, ke RT sukcesis, sed ĝi ne nepre rilatas al la kvalito de la cDNA-fadeno. Ĉar la internaj referencaj fragmentoj ĝenerale estas malgrandaj kaj esprimoplenaj, ili pli facile sukcesas pri inversa transskribo. Tamen la grandeco kaj esprimo de cela geno varias de geno al geno. La cDNA-kvalito ne povas esti taksata nur per interna referenco precipe por la celaj fragmentoj pli longaj ol 2 kb.
Iuj specimenoj havas kompleksajn duarangajn strukturojn, aŭ havas riĉan GC-enhavon, aŭ estas altvaloraj kun malmulta abundo. En ĉi tiuj kazoj taŭga inversa transkriptazo devas esti elektita laŭ la grandeco de la cela fragmento kaj la specimeno. Por RNA-ŝablonoj kun alta GC-enhavo kaj kompleksa duaranga strukturo, estas malfacile malfermi la duarangan strukturon je malalta temperaturo, aŭ kun komuna inversa transkriptazo. Por ĉi tiuj ŝablonoj, Quant Reverse Transcriptase povas esti elektita, ĉar ĝia inversa transskriba agado evidente pli bonas ol tiu de M-MLV-seria inversa transkriptazo, kiu povas inversigi transskribi diversajn RNA-ŝablonojn efike kaj transskribi RNA en cDNA-unuan fadenon laŭ la maksimuma mezuro. Kiam vi uzas ĝeneralan inversan transkriptazan kiton, 20 μl-sistemo povas efike inversigi transskribi 1 μg de totala RNA. Bonvolu atenti la maksimuman RT-kapaciton de ilaro. Se la ŝablono aldoniĝas tro, inversa transskribo favoros la RNA kun granda abundo. Tial estas pli bone ne superi la maksimuman kapablon de la sistemo.
A-1 Determini ĉu RNA estas grave degradita kaj se RT sukcesas
Ĝenerale, la kialo de la fiasko de interna referenca plifortigo ofte estas kaŭzita de grava RNA-degradado. Alia ebla kialo estas inversa transskriba fiasko. Interna referenco ne povas esti uzata kiel normo por juĝi la kvaliton de unu-fadeno de cDNA, sed ĝi povas esti uzata kiel normo por juĝi ĉu inversa transskribo sukcesas se ne ekzistas problemo pri la RNA-kvalito. La plej grava afero en la inversa transskriba procezo estas konservi konstantan temperaturon kaj konstantan reakcian sistemon por plibonigi la reakcian efikecon.
A-2 Determini ĉu la enkondukoj por plifortigi internajn referencajn genojn estas fidindaj kaj ĉu estas iuj problemoj kun reakciiloj uzataj en PCR.
Por relativa kvantigado, RNA devas esti kvantigita antaŭ inversa transskribo, kiu ankaŭ estas postulata en multaj inversaj transskribaj ilaroj, ekzemple, kvantigas la RNA-enigaĵon kiel 1 μg. Ĉar la inversa transskribita cDNA estas miksita solvo, inkluzive de RNA, oligo dT, enzimo, dNTP, kaj eĉ iom da DNA-restaĵo, devio estos kaŭzita, do estas maleble precize kvantigi la cDNA. Sekve necesas RNA-kvantigado. Konsiderante, ke la inversa transskriba efikeco samas inter diversaj specimenoj, la kvanto de cDNA akirita devas esti la sama, kaj la kvanta analizo povas montri la komparon de esprimaj niveloj de malsamaj genoj en la sama kvanto de totala RNA. Dum plenumado de relativa fluoreska kvanta PCR, kvanta cDNA eble ne necesas post inversa transskribo ĉar la interna referenca geno povas esti agata kiel referenco.
Ĝi ĉefe rilatas al la genoj, kaj inversa transskribo de longa fragmento ne estas farebla por plej multaj genoj. Unue, la efikeco de inversa transskribo estas multe pli malalta ol tiu de PCR. Due, la riĉa regiono de GC kaj duaranga strukturo de multaj genoj limigas ambaŭ inversan transskribon kaj PCR. Fine, la fideleco kaj plifortiga efikeco de PCR malfacilas samtempe garantii. En la procezo de inversa transskribo, neniu povas garantii akiri longan fragmenton por malaltaj kopiaj genoj, precipe per oligo dT. Koncerne 5 'UTR kun pli da GC, ĝi estas eĉ pli malfacila. Sekve, estas ankoraŭ racia metodo inversigi transskribaĵon kun hazardaj enkondukoj, trovi la naturajn intermamajn fendajn ejojn en la cela fragmento, plifortiĝi per segmentoj, kaj tiam plenumi la limigan digestadon kaj ligadon. Ĝenerale malfacilas rekte plifortigi fragmentojn pli grandajn ol 2 kb, sed ne ĉiam estas neeble akiri: 1. Unue garantiu la integrecon de RNA / mRNA, kaj TRIZOL-eltiro estas preferata. 2.M-MLV RT-PCR-ilaro povas esti rekte uzata. Plilongigu kalcinan tempon kaj pliigu ciklan nombron en la plifortiga procezo konvene. Alternative, nestita PCR povas esti aplikita, aŭ aranĝi unu aŭ du reagojn unue kun taŭge plilongigita denaturigo kaj etenda tempo antaŭ normala PCR-plifortigo, kiu povas helpi etendi fragmentojn. Atentu la fidelecon de la polimerazo. 3. Longa Taq povas esti uzata en PCR por akiri idealajn rezultojn. 4. Por apliko pri proteina esprimo, oni devas apliki altfidelan polimerazon.
Estas du specoj de inversa transkriptazo ofertita de TIANGEN: Quant / King RTase kaj TIANScript M-MLV. La ĉefa diferenco inter ili estas eniga kvanto de ŝablonoj. Kvanto estas unika inversa transkriptazo, kiu diferencas de la ofte uzata M-MLV derivita de la virina leŭkemio de Moloney. Quant estas nova altfika inversa transkriptazo rekombine esprimita per inĝenierio de Escherichia coli. Quant taŭgas por plifortigi 50 ng-2 μg de RNA kun alta inversa transskriba agado kaj alta rendimento. Kompare kun ordinara MMLV aŭ AMV, la plej granda karakterizaĵo de Quant estas, ke ĝi havas tre fortan afinecon kun RNA-ŝablonoj kaj povas inversigi transskribajn kompleksajn ŝablonojn sen alta temperatura denaturigo. Por ŝablonoj kun pli alta GC-enhavo, la inversa efikeco estas pli alta. Tamen ĉi tiu inversa transskribo havas RNase-H-agadon, kiu povas influi la longon de cDNA-produkto (taŭga por ŝablonoj <4,5 kb). Por konvencia inversa transskribo, TIANScript MMLV-inversa transskribado rekomendas. Ĉi tiu RTazo estas modifita enzimo kun tre malforta agado de RNasa H, kiu taŭgas por longa (> 5 kb) cDNA-sintezo.
Unupaŝa inversa transskribo kaj PCR-plifortigo estas kompletigitaj en la sama tubo sen malfermado de la tubkovrilo inter cDNA-sintezo kaj plifortigo, kio helpas redukti poluadon. Ĉar ĉiuj cDNA-specimenoj akiritaj estas uzataj por plifortigo, la sentemo estas pli alta, kun minimumo de 0,01 pg de totala RNA. Por sukcesa unupaŝa RTPCR, genospecifaj enkondukoj estas ĝenerale uzataj por iniciati cDNA-sintezon. La du-ŝtupa metodo, nome inversa transskribo kaj PCR-plifortigo efektivigas en du paŝoj. Unue inversa transskribo efektivigas de RNA-ŝablono por akiri cDNA, kaj la akirita cDNA estas submetita al unu aŭ pluraj malsamaj PCR-reagoj. La du-ŝtupa metodo povas uzi oligon (dT) aŭ hazardajn enkondukojn por gvidi la sintezon de la unua fadeno de cDNA, kaj povas inversigi transskribi ĉiujn mRNA-informojn de specifa provaĵo.
Produktaj kategorioj
KIAL ELEKTU NIN
Ekde ĝia estiĝo, nia fabriko disvolvas unuajn mondklasajn produktojn konforme al la principo
de kvalito unue. Niaj produktoj akiris bonegan reputacion en la industrio kaj valoran fidon ĉe novaj kaj malnovaj klientoj ..








